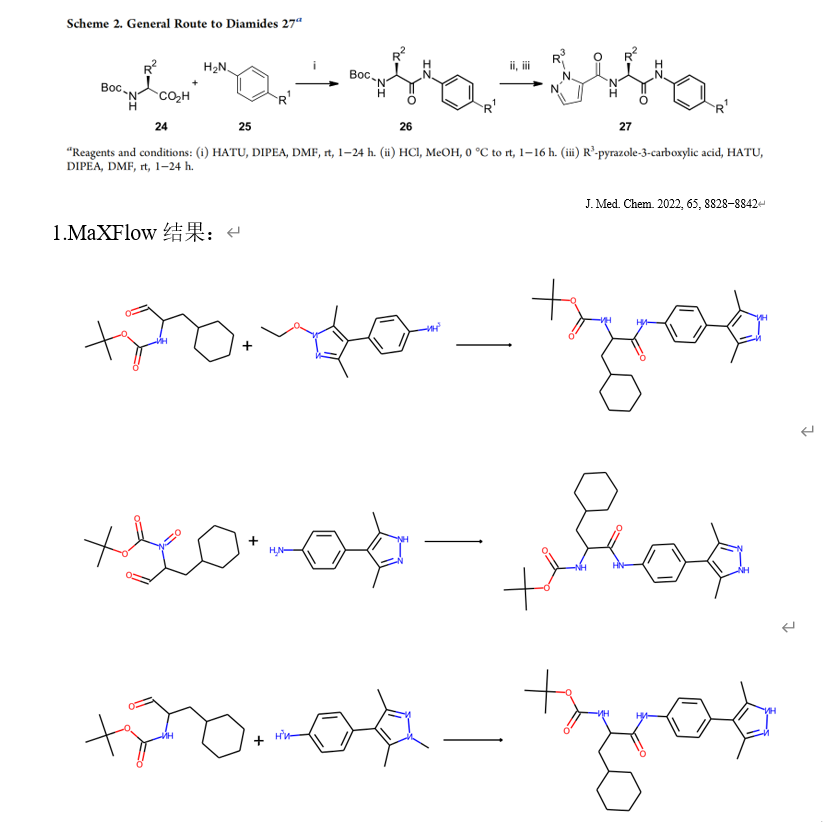
1. 机器学习或深度学习的训练使用数据量大概多少,预测精确度如何?是否会使用一些已知的数据进行测试?
答:针对不同的预测模型,我们采用权威的开源数据库进行训练,如对于逆合成路线预测,我们使用USPTO-50k数据库上的所有相关数据进行训练。对于精确度我们已经在尝试通过近期已报道的文献进行相关测评,如图所示为使用MaXFlow预测文献中化合物的合成路线与实际路线的对比,结果表明MaXFlow的预测结果与文献一致。
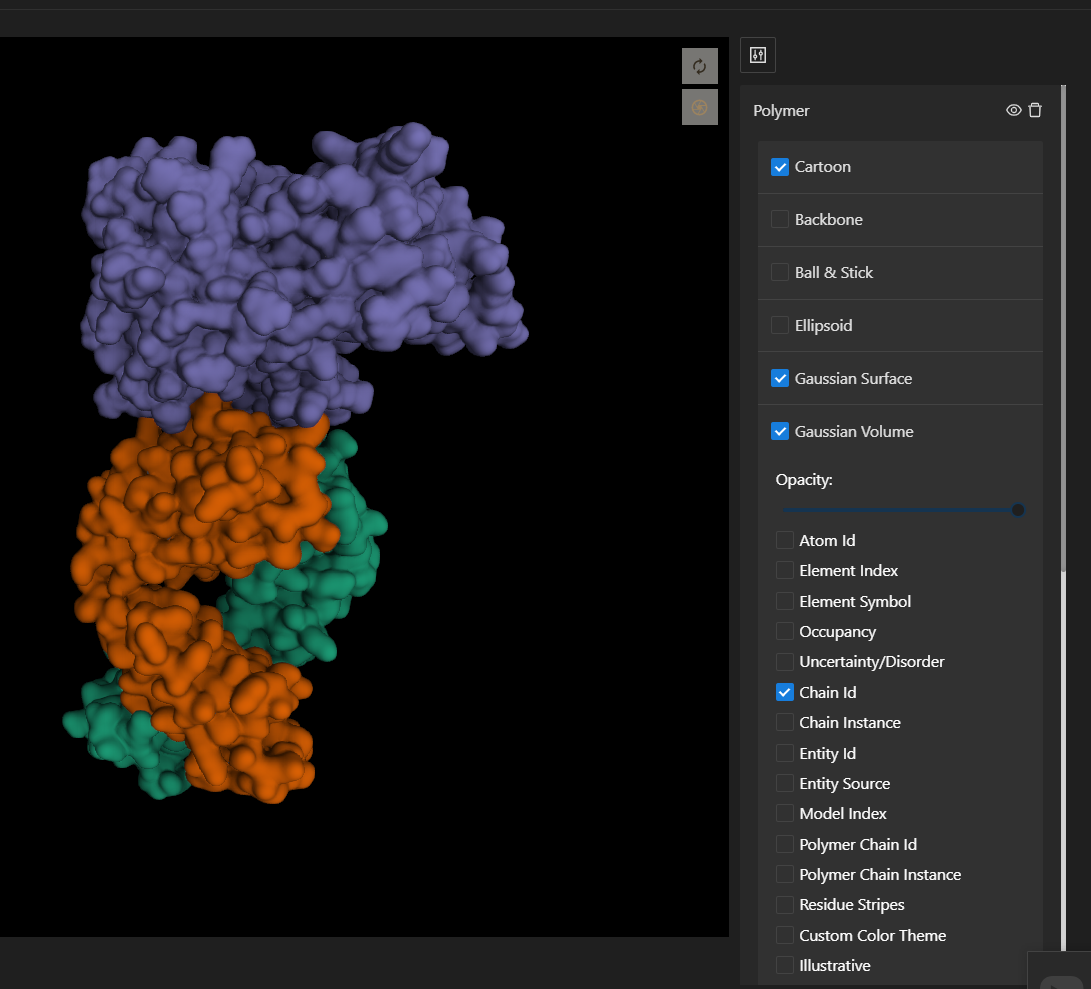
2. 抗原抗体对接是刚性对接吗,准确率如何?
答:目前MaXFlow是刚性对接,采用MEGADOCK引擎,其针对特殊的抗体结构会进行构象优化。同时MEGADOCK使用多种技术减少对接所需的计算时间,例如一种新的评分函数,称为真正的成对形状互补(rPSC)评分。研究表明完成对接计算的PPI筛选,MEGADOCK的穷举能力比传统对接软件ZDOCK快7.5倍,计算结果基本一致。
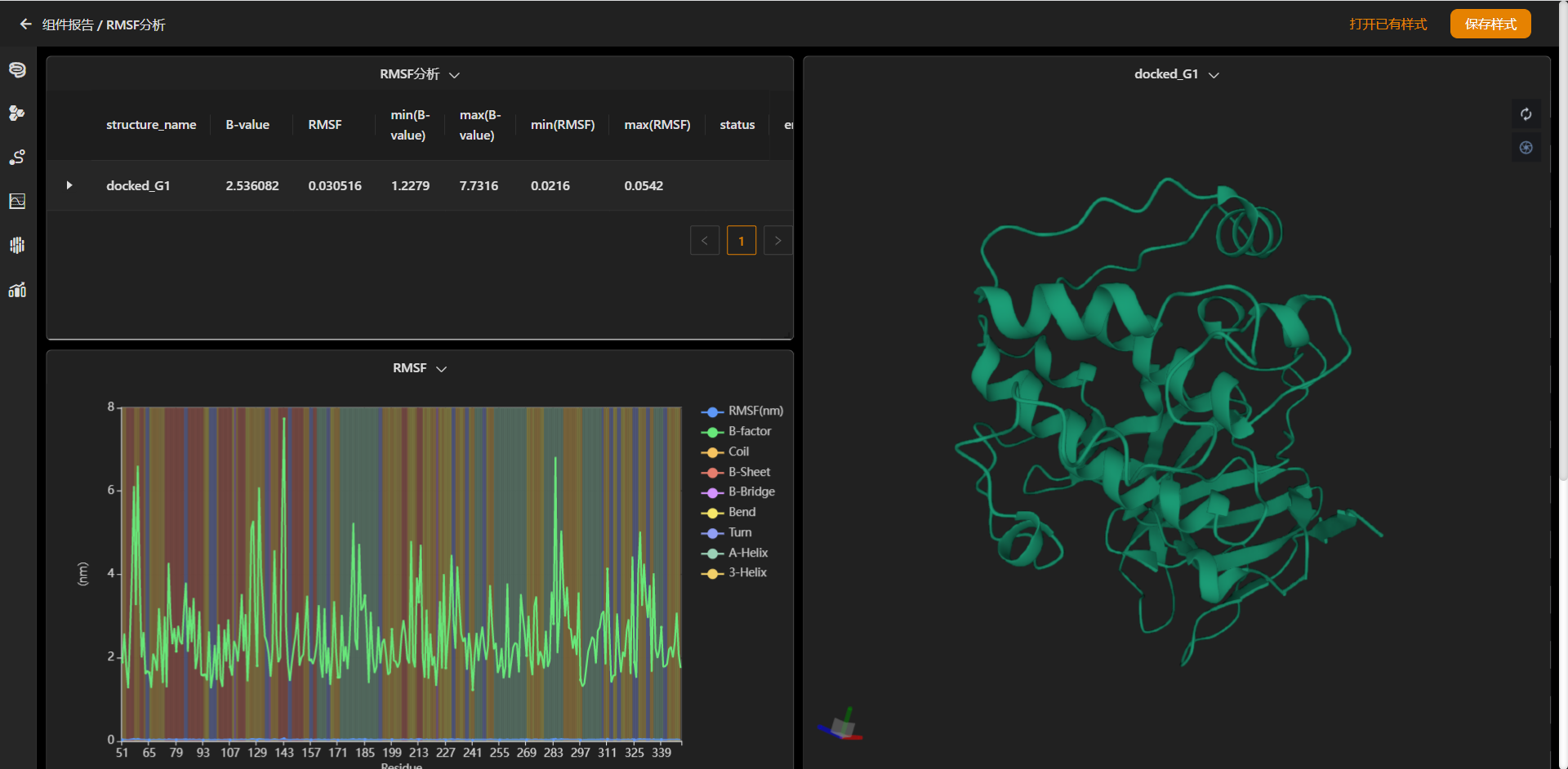
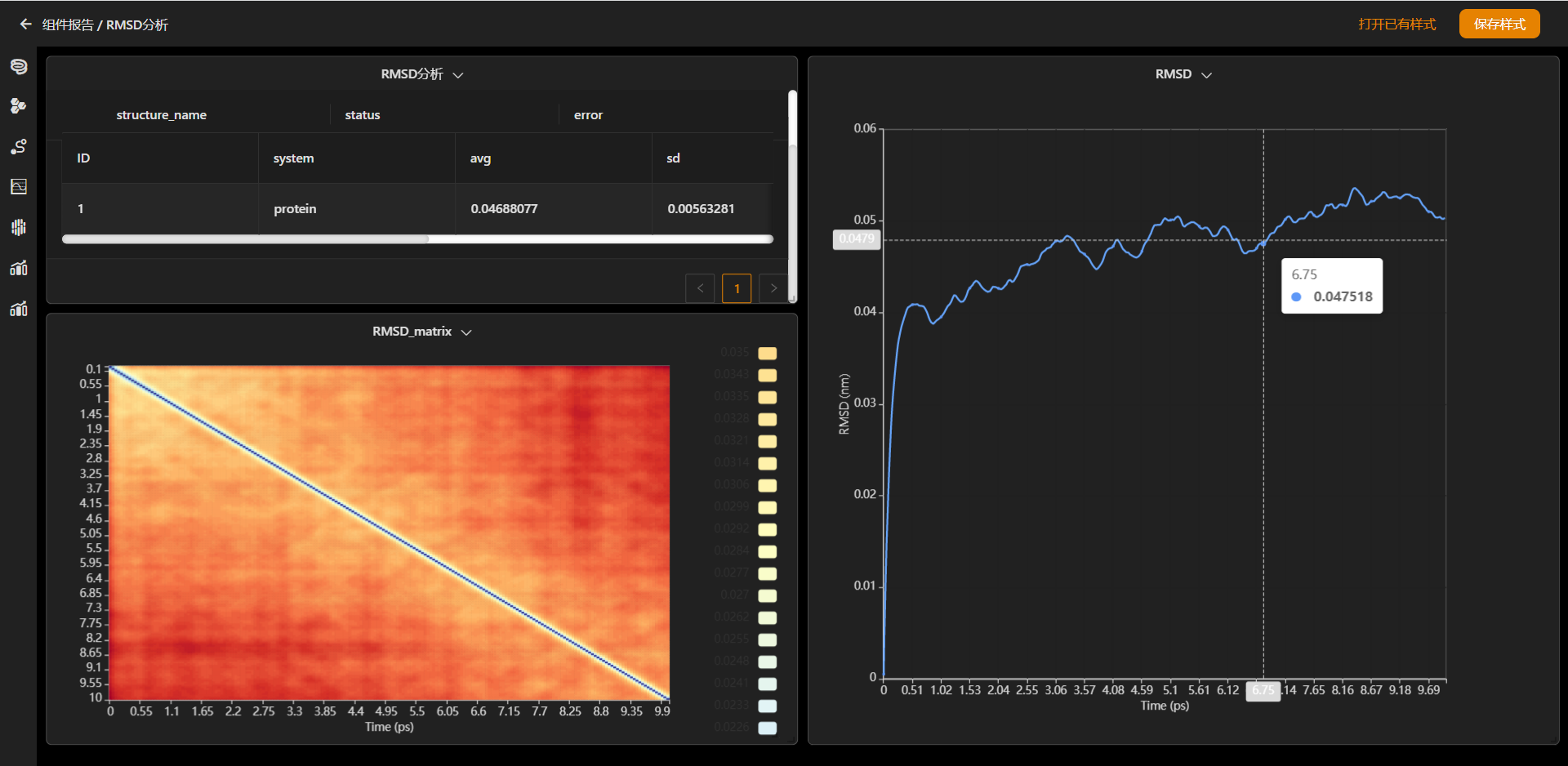
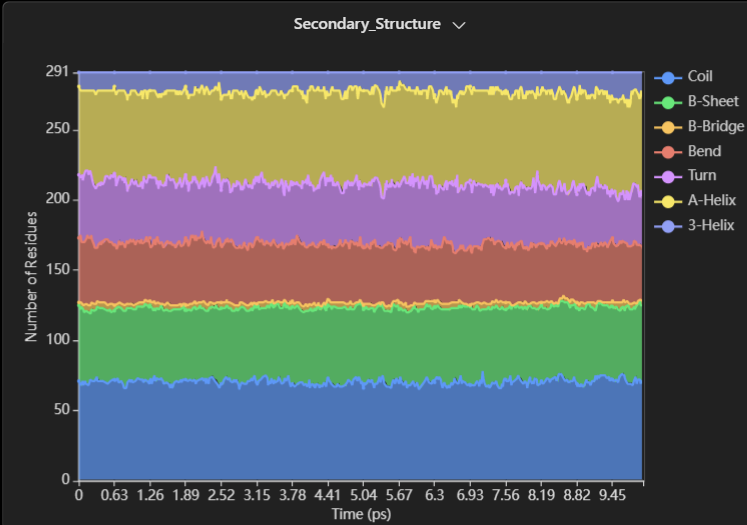
3. MaXFlow是否能够进行细致的相互作用分析?
答:目前动力学模拟部分不仅支持基础的理化性质分析、骨架波动分析(RMSD、RMSF),还将支持蛋白质二级结构分析、热点残基分析(丙氨酸扫描、能量分解)、结合自由能分析(FEP自由能、MMPBSA)、相互作用分析(氢键分析、盐桥分析)、构象转变分析等,可以从不同角度给出相互作用全面的分析。
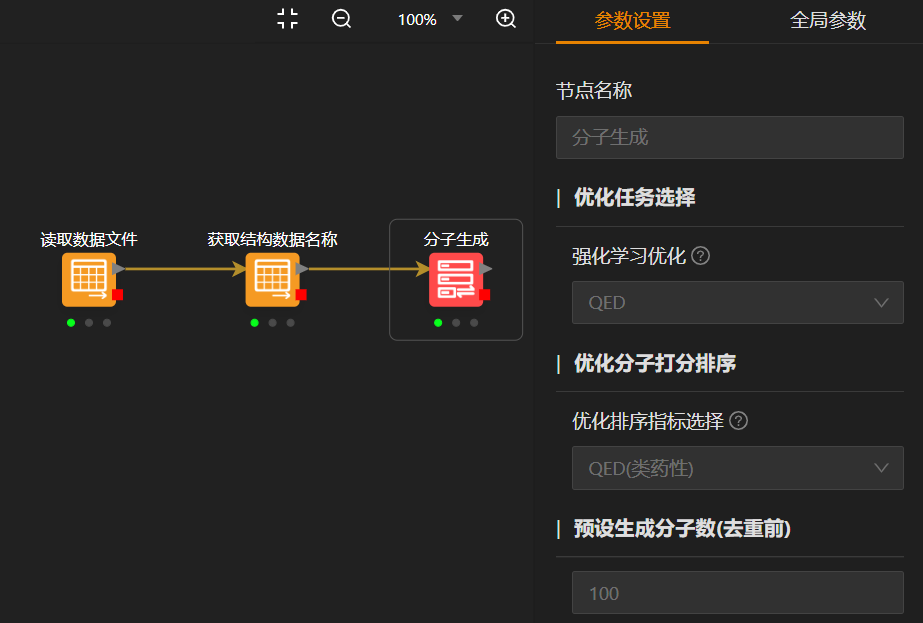
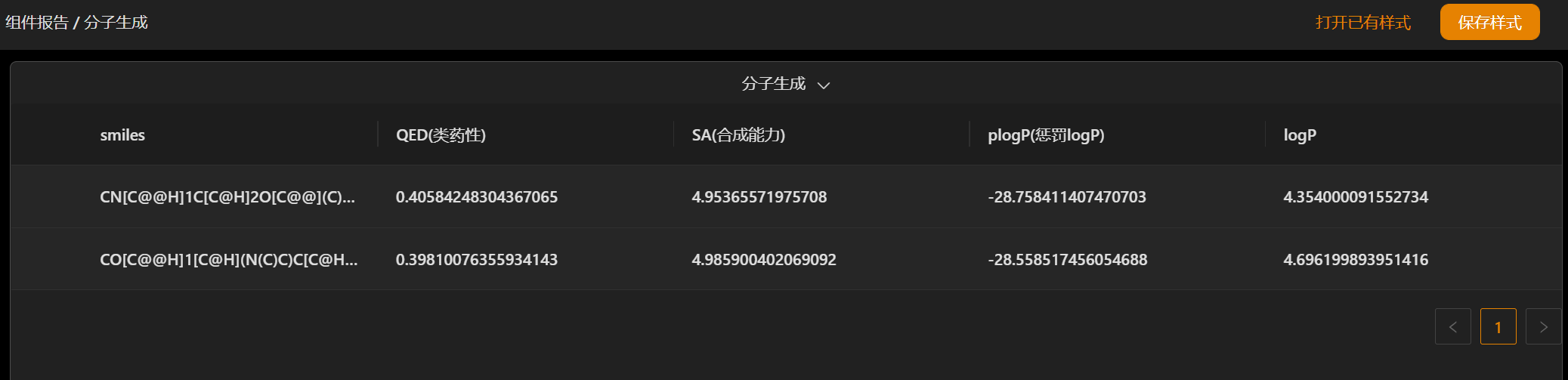
4. 新生成的分子模型,创新性有没有指标来评价,而且生成的分子数量很多,如何验证是否已经存在?成药性的打分标准是什么,仅仅按照(QED)来评价合理吗?
答:为了评估生成分子的新颖性,MaXFlow通过ISOMAP算法应用于从ZINC数据库获得的训练数据集,构建从指纹到二维空间的映射,灰点代表ZINC数据库中的分子,热图点代表生成分子随时间的分布,从而实现生成新颖分子的评估。成药性指标包括类药性QED、合成性分数SAscore、水溶性指标logP以及plogP等,进一步还可通过分子对接或者药靶亲和力进行分子评价。
5.重链和轻链是否可以在结构上标识CDR区?
答:抗体目前已经有了序列注释组件,包含IMGT、Kabat、Chothia和Martin四种可选编号系统对抗体进行注释,可以给序列标注CDR区。
6.CDR优化,组件里AI模型,可以直接实现优化吗?
答:CDR区的loop因为要结合多种抗原而会具有更多样的结构,导致此区域的建模准
确性更低,特别是第三个CDR loop(CDR-H3)。MaXFlow可以通过AI直接预测CDR区的loop并进行能量最小化获得更准确的CDR结构。