
这项研究借用GFC和RP-HPLC并结合感官评估和电子舌等方法或技术,从卵形鲳鲹水解产物分离和纯化出鲜味肽,并通过Nano-HPLC-MS/MS鉴定了5个肽,其氨基酸序列为APAP,ASEFFR,AEASALR,LGDVLVR,WDDMEK,阈值范围为0.034 - 0.306 mmol/L。通过分子对接来分析鲜味肽和受体之间的相互作用。结果表明,T1R3中Asp192和Glu301可能在鲜味肽的鲜味释放中起重要作用,并且氢键和静电相互作用是两者之间最重要的相互作用力。
同源建模(Homology modeling)是利用信息技术的手段,可以直接从蛋白的一级结构(氨基酸序列)预测蛋白质的高级结构(主要为三级结构)。人们可以通过使用一个或多个已知结构的蛋白(模板蛋白,template)来构建未知结构蛋白(目标蛋白,target)的空间结构。BIOVIA Discovery Studio为用户提供了一整套利用Homology modeling方法自动预测蛋白质空间结构的工具。用户只需要提供蛋白质的氨基酸序列既可以轻松完成同源模型的构建及模型可信度评估的工作。
分子对接(Molecular docking)是基于结构药物设计的核心模拟手段,依据受体与配体作用时的几何匹配和能量匹配过程,模拟受体-配体相互作用,预测两者间最佳的结合模式和结合亲和力。采用分子对接模拟技术,科研人员可以进行基于结构的药物虚拟筛选,药物分子的结构改造,配体和受体相互作用的机理研究等工作,从而大大提高实验效率。在BIOVIA Discovery Studio这一分子模拟的综合平台中,分子对接程序包含Libdock、CDOCKER、Flexible Docking,这三种对接算法各有优势,能够满足广大科研工作者的多种应用需求,为其提供配体受体间相互识别的“利器”。
ref:LWT - Food Science and Technology. Accepted 8 January 2021, IF=4.006
链接:https://doi.org/10.1016/j.lwt.2021.110887
一、研究背景
鲜味通常被称为继酸味,甜味,苦味和咸味之后的第五种基本口味,是人类最重要的食品接受口味之一。天然存在于各种肉类,奶酪,海鲜和蔬菜中的许多物质,例如一些游离的L-氨基酸,核苷酸,双功能酸,肽及其衍生物或反应产物,可能会诱发类似味精的现象。由植物或动物蛋白酶水解和发酵产生的许多肽成分(主要包括二肽和三肽)已被证明具有浓厚的鲜味,许多肽是由谷氨酸和亲水性氨基酸残基组成,具有类似于谷氨酸钠的味觉特性。这些具有复杂口味特征的鲜味肽在食品风味的形成中起着重要作用,可改善食品质量,增强口感并增加消费者的食欲。由于鲜味肽对食品营养和生物活性有很大的影响,因此可以开发成天然、营养和安全的食品成分,从而获得高质量的产品。
鲜味,甜味和苦味是由促味剂与G蛋白偶联受体(GPCR)家族结合而引发的,包括mGluR1,mGluR4和T1RI/T1R3。近年来,分子模拟的方法如同源建模和分子对接已被用来揭示鲜味肽与受体T1R1/T1R3之间的相互作用。但是,不同的鲜味肽可能具有不同的结合位点,这是用于确认或预测味觉产生的必要辅助因子。
二、研究过程
在这项研究中,结合感官评估和电子舌技术,利用GFC和RP-HPLC分离和纯化了来自卵形鲳鲹水解产物的鲜味肽或鲜味增强肽,并通过Nano-HPLC-MS/MS鉴定了5个肽,其氨基酸序列为APAP,ASEFFR,AEASALR,LGDVLVR,WDDMEK,然后进行合成以进行感官评估。
为进一步验证它们是否是鲜味肽,先通过借用BIOVIA Discovery Studio 中homology model构建出了鲜味味觉受体T1R1/T1R3的三维结构,并对结构采用Ramachandran plot进行了验证。结果显示97.72%的氨基酸残基在允许区域内,2.28%的残基在不允许区域内,这表明该模型具有很高的可靠性(图1)。然后将鉴定出的肽段再用CDOCKER进行分子对接研究鲜味肽段与味觉受体T1R1/T1R3之间的相互作用机制。图2展示了最佳的对接模式,具有最小的对接能量。结果表明,经鉴定的5个肽段都可以对接于T1R3腔VFTD中的结合口袋。但是不同鉴定的肽的对接结果是不同的。T1R3的结合位点主要包含Glu301,Asn150,Ser172,Asp192,Asp219,Ser275,Ser276,Arg277,His326,Gln327,Lys328,Asn450,Lys335。该研究表明,Glu301,Asp192,Asn150,Asp219在已鉴定的鲜味肽与T1R3的相互作用中起着至关重要的作用。另外,Ser例如Ser148,Ser172,Ser275,Ser276,Ser384和Ser385,可能在鲜味肽与T1R1/TIR3的结合中起到辅助作用。结合位点的结果可以为未知肽的准确预测提供有价值的依据。并且对接结果表明,常规的氢键和静电相互作用是结合中的主要作用力。因此,通过分子对接有助于我们更好地理解鲜味肽的机理。
三、结论
在这项研究中,通过GFC和RP-HPLC并结合感官评估和电子舌等方法或技术以及连续分离进行鉴定,从卵形鲳鲹水解产物分离和纯化出了5种新颖的鲜味肽,鲜味和咸味增强肽。并使用分子对接的方法来解释肽与受体间的作用机制,结果表明,鉴定的肽WDDMEK和LGDVLVR可以引起鲜味和鲜味增强,并且引起鲜味和鲜味的增强主要是肽与受体间形成了常规的氢键和静电相互作用。因此,这些研究结果可为我们更好地了解卵形鲳鲹的鲜味机理提供依据,为进一步开发海洋资源鲜味肽提供一种有效实用的方法。
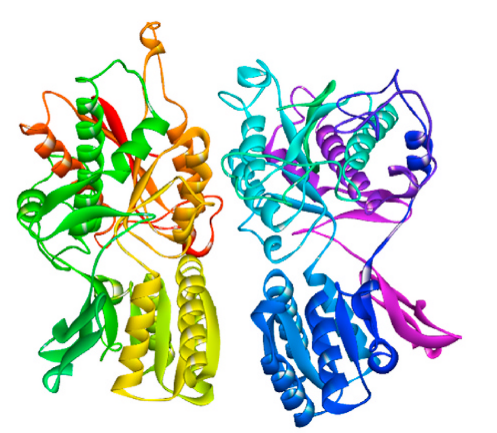
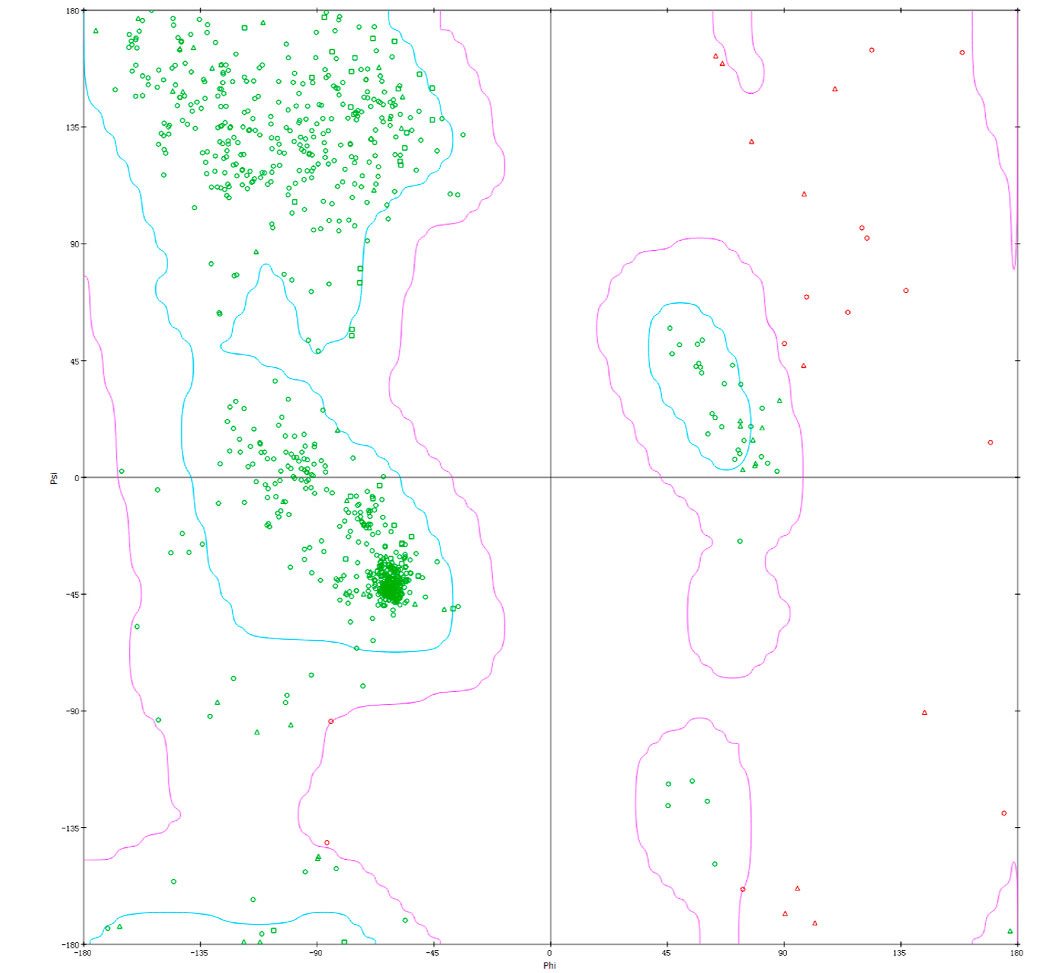
图1 (a)鲜味受体T1R1/T1R3的结构;(b) T1R1/T1R3同源模型的验证
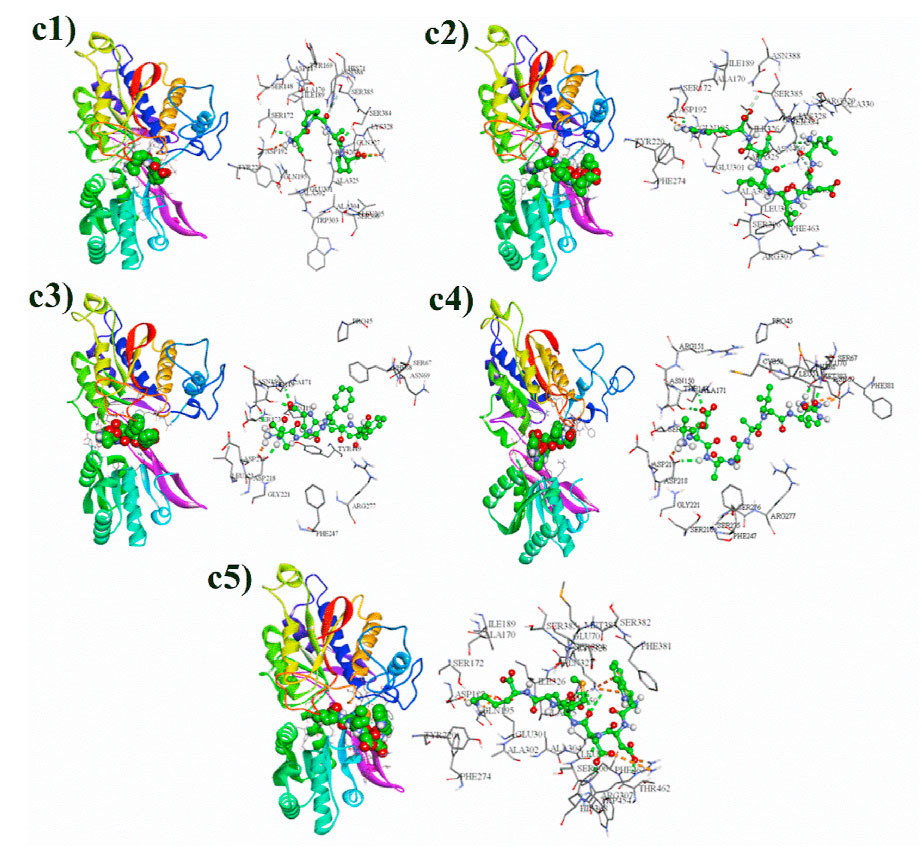
图2 T1R1/T1R3受体与5个多肽的分子对接结果
(c1) APAP;(c2) LGDVLVR;(c3) ASEFFR;(c4) AEASALR;(c5) WDDMEK。
为什么选择 BIOVIA Discovery Studio?
1. BIOVIA Discovery Studio 中共有三种对接程序,这三种对接算法各有优势,能够满足广大科研工作者的多种应用需求;
2. BIOVIA Discovery Studio 中有多种对接结果的评价分析手段,包括2D/3D相互作用图分析(可以分析氢键、疏水作用、卤键等多种非键相互作用类型)、结合口袋表面分析、氢键热图、多种打分函数、结合能的计算等;
3. BIOVIA Discovery Studio 提供一整套同源建模的工具,使用DS,可以轻松完成所有的同源建模过程,模板识别、序列比对、自动建模、模型的评估与优化;
4. BIOVIA Discovery Studio 中同源建模工具可以预测多种类型生物大分子的结构,包括:球蛋白、抗体、跨膜蛋白等;
5. BIOVIA Discovery Studio 中同源建模的核心程序是MODELER,该算法非常经典,引用率比较高。
6. BIOVIA Discovery Studio 应用广泛,操作简便,图形化界面十分友好,结果易于分析。