
目前前列腺癌的检测和化疗治疗方法有限。前列腺特异性膜抗原(PSMA)在前列腺癌中过表达,可以作为成像和给药的靶标。我们开发并鉴定了四种纳米抗体,它们在PSMA过表达的细胞中具有特异性的结合和内化,并特异性地在PSMA +肿瘤细胞中积累。然后我们将其中一个纳米抗体与细胞毒性药物阿霉素偶联,证明了该偶联物可以特异性地进入PSMA +细胞中,并在细胞内释放药物,进而诱导细胞毒性。
为研究该纳米抗体(NB7)与PSMA的作用机制,采用 Discovery Studio 中的ZDOCK,ZRNK以及RDOCK程序将NB7与PSMA进行了对接,发现NB7与PSMA一个单体主要通过CDR3和CDR1发生相互作用,而CDR2和几个非CDR残基第二个单体发生相互作用。进一步的体内研究表明,当用商用阿霉素和低42倍剂量的纳米抗体偶联阿霉素治疗小鼠时,肿瘤生长抑制的程度是相似的,这证明了纳米抗体偶联药物的有效性。这些数据表明了纳米抗体在前列腺癌成像和化疗药物靶向输送方面的前景。
蛋白对接(ZDOCK/RDOCK)是ZDOCK是一种基于快速傅里叶转化相关性技术的刚性蛋白对接算法。算法中快速傅里叶转化相关性技术被用于搜索蛋白-蛋白系统的平动和转动空间。RDOCK是一种基于CHARMm的能量优化过程,用于优化ZDOCK所寻找到的蛋白-蛋白复合物的结合构型,并使用能量打分函数给这些结合构型打分。
ref:J. Med. Chem, IF= 6.054
链接:https://dx.doi.org/10.1021/acs.jmedchem.0c00418
前列腺癌(PCa)通常是通过抗体结合前列腺特异性抗原(PSA)测血清浓度的方法来检测,但这种检测方法有较高的错误率。此外,虽然化疗经常用于治疗去势耐药的前列腺癌,但一些潜在有效的针对PCa的化疗,如阿霉素,在肿瘤细胞内蓄积不足,分布范围太大,导致治疗效果低,非特异性毒性高。因此,迫切需要新的方法来检测PCa和靶向给药细胞毒性药物。一个有希望解决这两个问题的靶点是前列腺特异性膜抗原(PSMA); 一种在PCa中过表达的跨膜蛋白,可能是由于其叶酸水解酶活性,导致细胞增殖。
到目前为止,大多数被发现与PSMA细胞外区域有足够高亲和力(纳米范围)的蛋白是单克隆抗体或抗体片段。由于血清半衰期较长,抗体的生物分布广泛,往往会降低其在循环中的信噪比,并使其长期维持在循环中。研究发现,当抗体偶联于细胞毒性放射性同位素时,这些效应会增加毒副作用;当抗体偶联于药物时,这些效应会降低特异性,因为抗体药物偶联物可能内化到非肿瘤细胞。此外,大尺寸的抗体常常阻碍它们穿透异常肿瘤组织核心的能力,从而极大地降低了它们的药物传递效率,抗体片段可能解决这些问题,但它们往往表现出较弱的结合和低稳定性,并可能暴露之前被掩蔽的免疫抗原表位。纳米抗体则表现出很好的亲和力。
在本研究中,我们从注射了重组人PSMA细胞外区域的骆驼血清中分离并选择了4个NBs。然后,我们评估了这些NBs的结构、PSMA结合表位、亲和力特异性,同时借助Discovery Studio中的ZDOCK,ZRNK以及RDOCK程序将NB7与PSMA进行了对接以研究作用机制,发现NB7与PSMA一个单体主要通过CDR3和CDR1发生相互作用,而CDR2和几个非CDR残基第二个单体发生相互作用,并将最有效的NB7与DOX偶联,从而生成一种新的NB药物偶联物,可以特异性靶向PSMA表达细胞。我们的研究结果表明了使用基于纳米抗体的偶联药物在诊断和治疗前列腺癌方面的潜力。
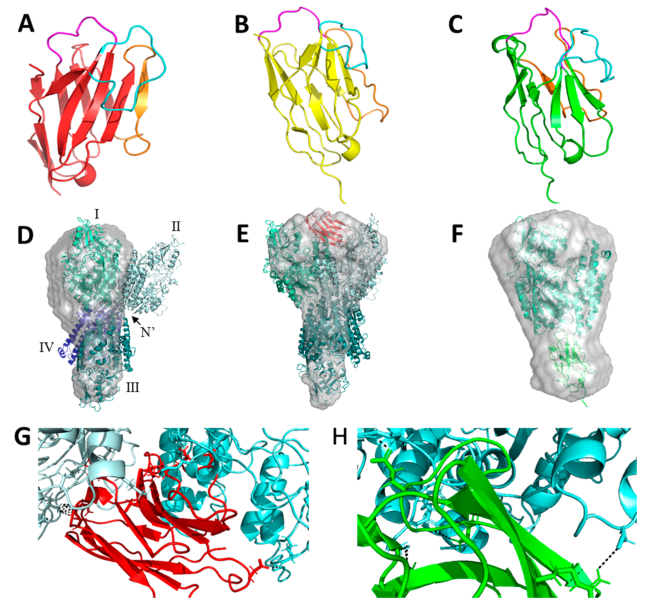
Figure. Structural analysis of NBs and their PSMA-binding epitopes. (A−C) Solved crystal structures of NB7 (A, red; 2.65 Å, PDB code 6XXN), NB8 (B, yellow; 1.5 Å, PDB code 6XXO), and NB37 (C, green; 1.5 Å, PDB code 6XXP). CDRs 1, 2, and 3 are labeled in magenta, orange, and cyan, respectively. (D−F) Structural reconstruction of protein complexes, based on their SAXS-resolved low-resolution structures (gray mesh), fitted with the crystal structure of PSMA (0.5 mg/mL), either alone (D; PDB code 1Z8L) or with 0.2 mg/mL NB7 (E, red; PDB code 6XXN) or NB37 (F, green; PDB code 6XXP). The PSMA monomers are labeled individually by Roman numerals; biological dimers are formed by I + II and III + IV, while nonbiological dimers are formed by I + III and II + IV. (G−H) Computational docking analyses of PSMA and NB7 (G) and of PSMA and NB37 (H). (G) PSMA monomers are colored in light blue and cyan, while NB7 is colored in red. Key interactions are shown as black dashed lines. (H) PSMA is colored in cyan, and NB37 is colored in green. Key interactions are shown as black dashed lines.
为什么选择Discovery Studio?
1. Discovery Studio 中ZDOCK是一种基于快速傅里叶转化相关性技术的刚性蛋白对接算法。
2. Discovery Studio中ZDOCK算法中快速傅里叶转化相关性技术被用于搜索蛋白-蛋白系统的平动和转动空间。
3. Discovery Studio中RDOCK是一种基于CHARMm的能量优化过程,用于优化ZDOCK所寻找到的蛋白-蛋白复合物的结合构型,并使用能量打分函数给这些结合构型打分。