
研究背景
结直肠癌是一种常见的恶性肿瘤,与腺瘤性息肉病和Wnt信号通路的异常活化有关。微管蛋白是细胞骨架的重要组成部分,参与细胞分裂、运输、信号转导等多种生理过程。微管蛋白的动态不平衡是维持其功能的关键,而一些能够干扰微管动态不平衡的小分子药物,如紫杉醇、长春新碱、秋水仙碱等,具有抗肿瘤活性,能够阻止细胞有丝分裂,诱导细胞凋亡。这些小分子药物主要靶向微管蛋白的三个位点:紫杉醇位点、长春新碱位点和秋水仙碱位点。其中,秋水仙碱位点位于β-微管亚基的N端,靠近α-β二聚体的界面。秋水仙碱位点结合的小分子药物能够抑制微管聚合,降低微管稳定性,从而抑制肿瘤细胞增殖和转移。
研究目的
目前,已有一些秋水仙碱位点结合的小分子药物,如ABT-751、CKD-516等。然而,这些药物仍然存在一些缺陷,如毒副作用大、耐药性高、选择性低等。因此,寻找新型的、高效的、安全的秋水仙碱位点结合的小分子药物是一个迫切的需求。
案例文献作者利用机器学习算法,从大规模的化合物库中筛选出了一些具有高亲和力和选择性的候选分子,并对其进行了分子对接模拟,以揭示其与靶标蛋白的结合模式。然后,作者对这些候选分子进行了一系列实验实验。结果表明,这些分子能够与微管蛋白的秋水仙碱位点结合,有效抑制了微管聚合,使细胞周期停滞在G2/M期,诱导了细胞凋亡,并表现出强效的体外抗转移活性。这些分子为开发新型的结直肠癌治疗药物提供了有价值的参考。
AIDD+CADD虚拟筛选-复现内容和结果
本文使用MaXFlow平台实现了AIDD+CADD虚拟筛选过程中所需的各项计算任务。用户可以通过简单拖拽组件,使用已有活性的小分子数据库和默认参数,构建出准确率84.01%的随机森林分类模型(准确率与文献的85.12%一致;MaXFlow中可通过数据预处理、特征工程和超参数优化进一步提升模型性能),并用于第一轮虚拟筛选。筛选后结构,可以使用MaXFlow中行业标准的分子对接程序进行第二轮虚拟筛选,并获得结合模式和亲和力预测结果。
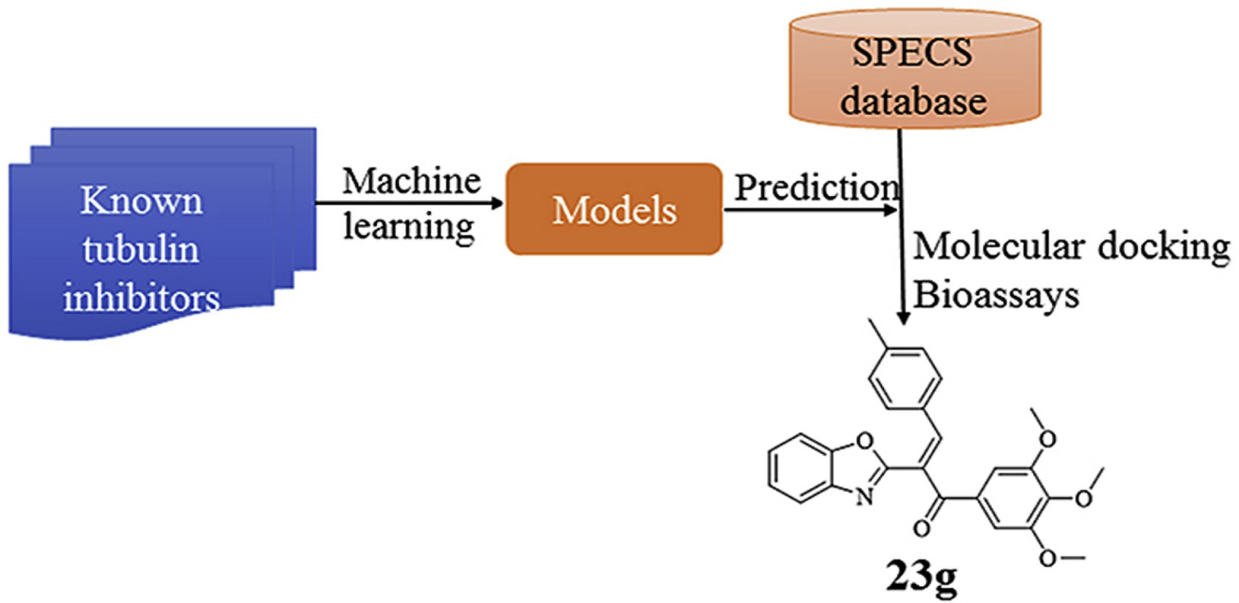
图1.AIDD+CADD虚拟筛选案例
(Guo Q, Zhang H, Deng Y, et al. Ligand-and structural-based discovery of potential small molecules that target the colchicine site of tubulin for cancer treatment. European Journal of Medicinal Chemistry, 2020, 196: 112328.)
数据准备
从文献中获得QSAR模型的训练数据,构建成包含化学结构SMILES式和分类标签的表格形式(https://doi.org/10.1016/j.ejmech.2020.112328)。模型预测使用线上获取的SPECS数据库(https://www.specs.net/)
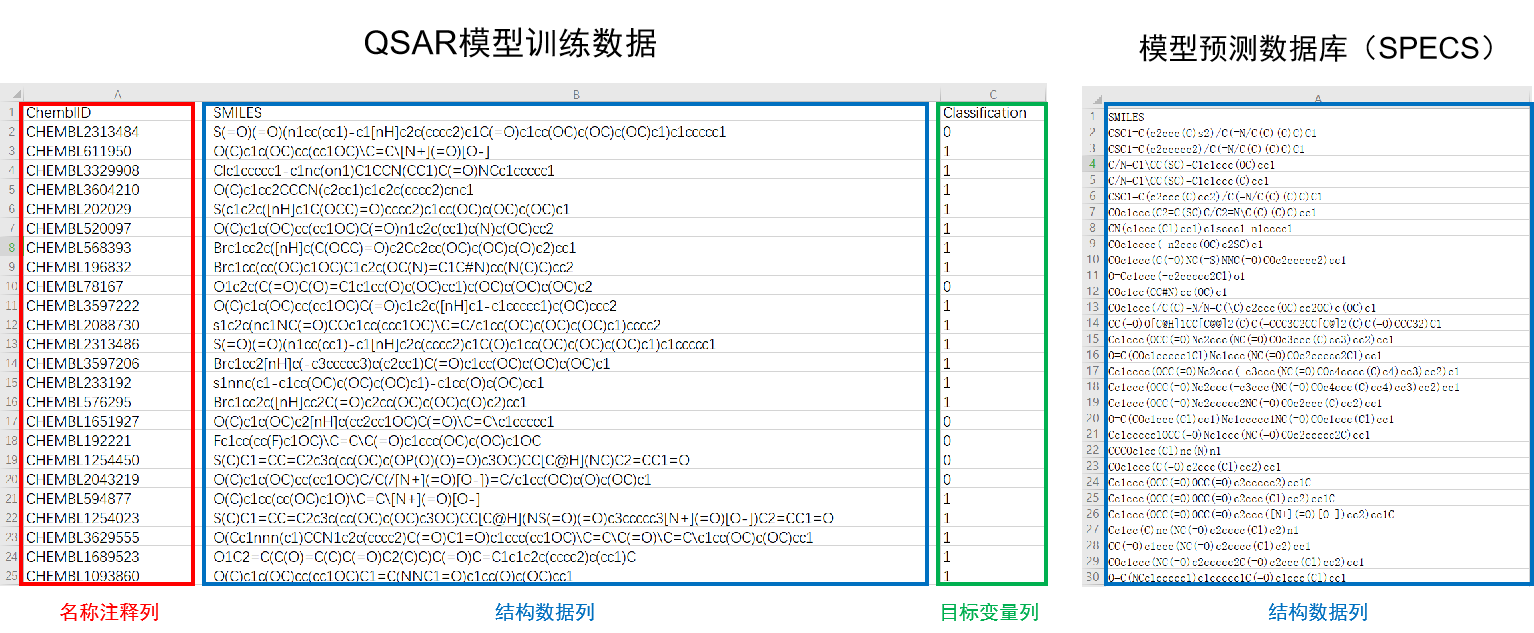
图2. QSAR模型训练数据和预测数据
机器学习QSAR模型
在MaXFlow工作流构建页面,构建出机器学习工作流,将模型训练数据导入“读取数据文件”组件,使用“设置注释列”注释不参与模型构建的化合物名称,使用“获取结构数据名称”获取SMILES式表头,使用“目标变量y”指定分类指标表头。接着使用“分子指纹计算”组件,根据输入化合物的SMILES式,计算获得一系列化合物描述符,用于模型构建。使用“数据集划分”组件划分参与模型构建的训练集和用于评估模型的测试集。使用“互信息过滤”组件,计算特征与标签之间的互信息值并进行筛选,使得数据能更好表达问题的本质。使用“随机森林分类建模”获得模型,并用“分类型模型评估”组件获得模型评估结果。
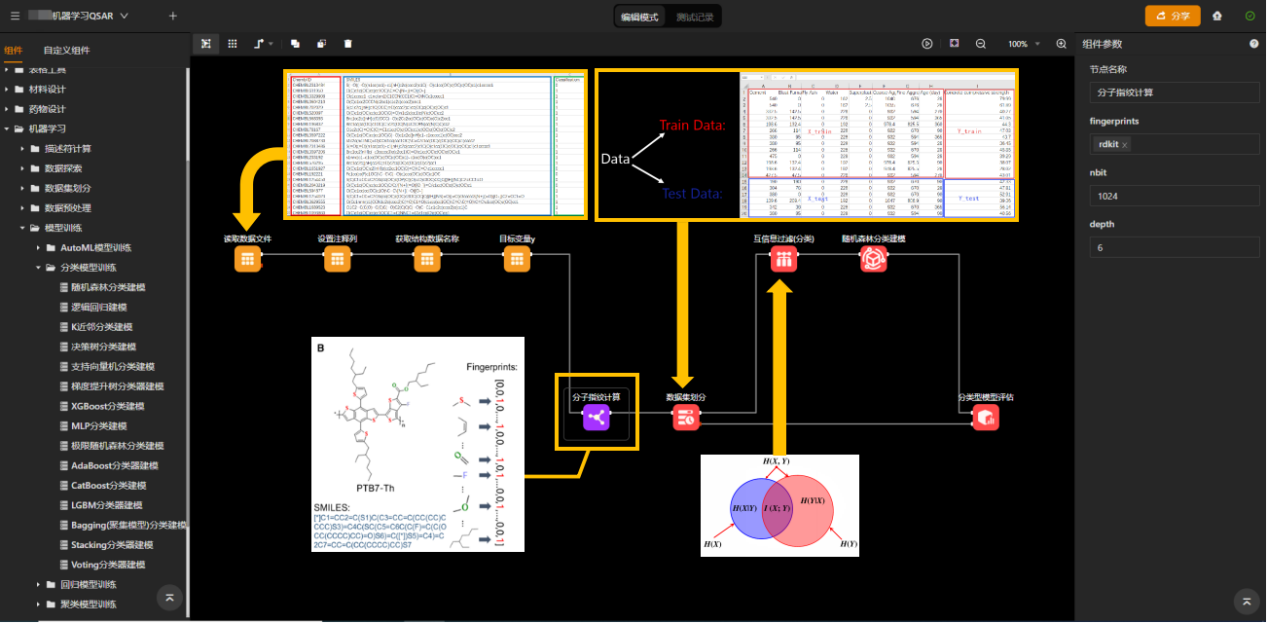
图3. QSAR模型构建工作流
模型评估结果显示,使用默认参数,未经过超参数优化的机器学习QSAR模型准确度即可达到84.01%与文献相当;95.03%的召回率说明该模型可以假阴性结果较少,可以经可能保留活性结构用于后续筛选;86.78%的AUC表明该模型有较强的分类能力。
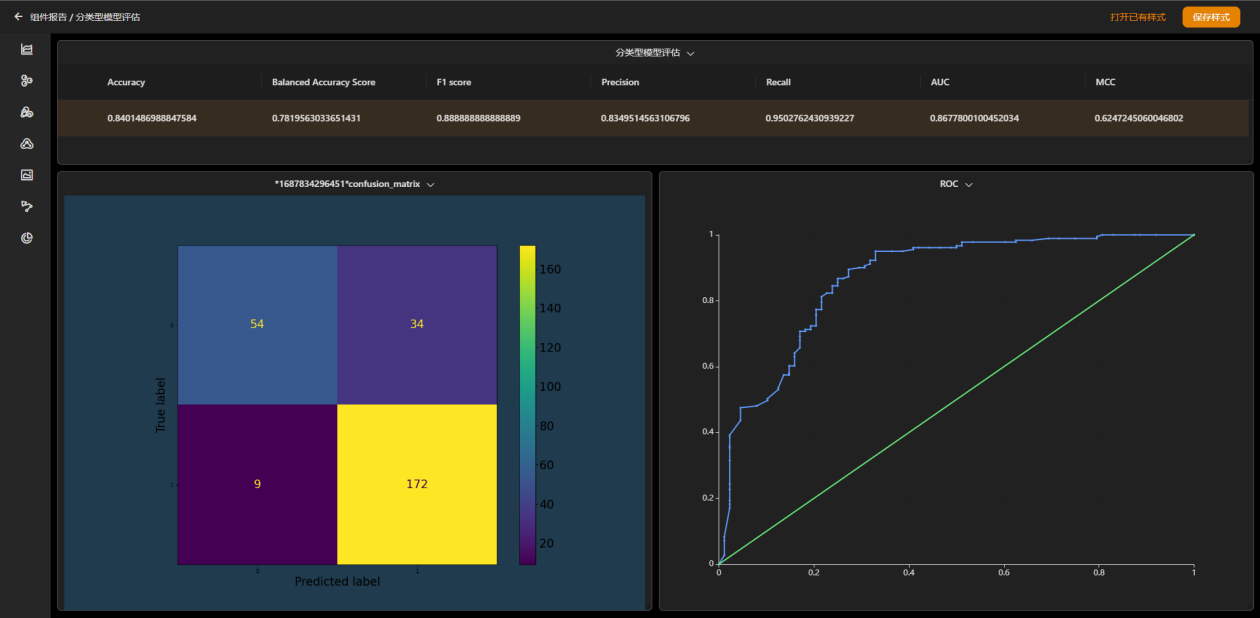
图4.模型评估结果

图5.文献截图和结果
构建完成的QSAR模型可以通过简单的工作流用于活性预测,实现虚拟筛选的目的
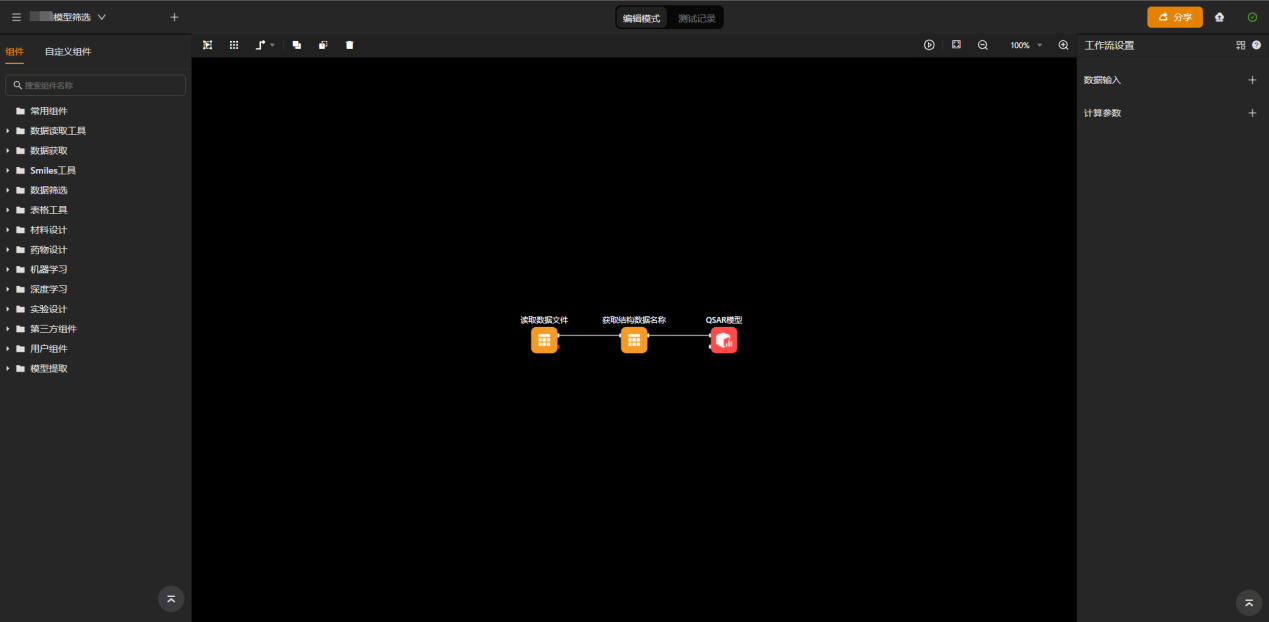
图6、机器学习QSAR模型虚拟筛选工作流
根据构建分类或回归模型,在计算结果的组件报告处可获得预测结果和分子三维结构
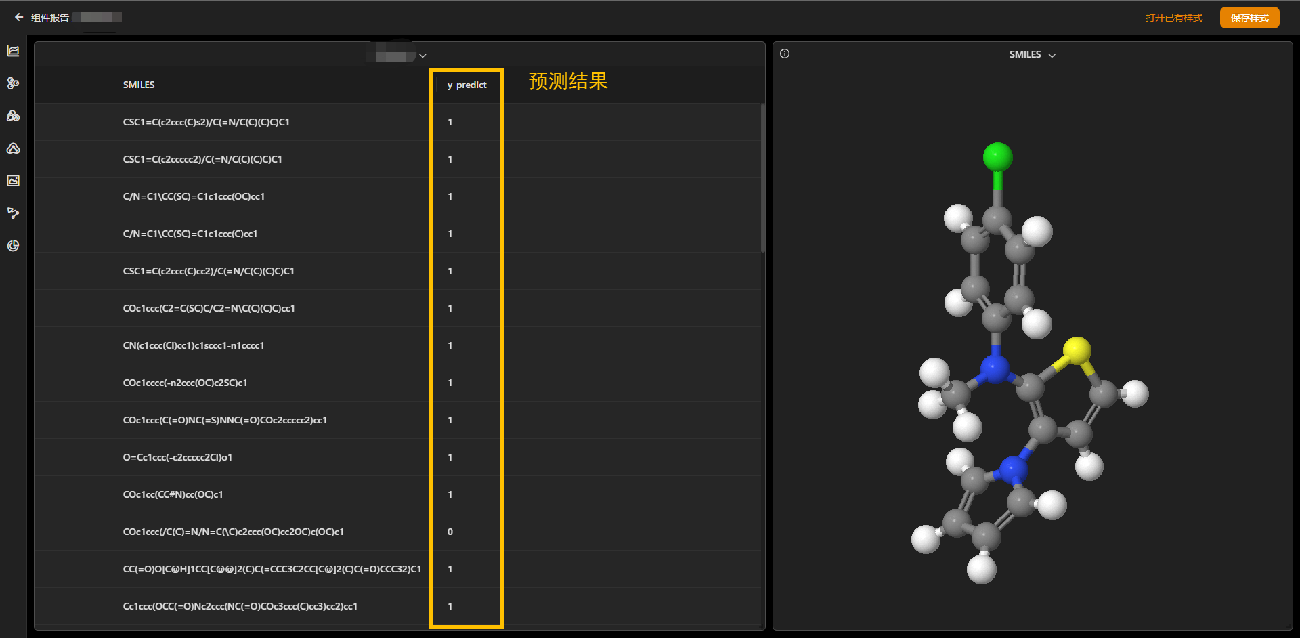
图7、机器学习QSAR模型虚拟筛选结果
训练完成的各类型机器学习模型以及其他计算流程均可封装成APP。允许将分享至组织内,供其他成员下载。使用时只需要上传输入文件,即可通过固化的流程计算获得一系列预测的数值。

图8、机器学习模型APP使用示意
分子对接
机器学习QSAR模型虚拟筛选过后的分子可使用分子对接进行第二轮虚拟筛选,通过打分函数的结合亲和力预测结果,进一步获得理论上活性更好的分子结构。首先通过PDB数据库获取蛋白质晶体结构,通过“大分子预处理”删除非标准残基并只保留两条需要计算的链,提高计算效率。在“分子对接”组件中,根据晶体中的配体结构,设置对接的网格参数。在另一条分支上,读入筛选后的分子表格,使用“SMILES转3D”组件转换出用于分子对接的小分子结构。将组件如图所示相连,即可提交任务,实现基于结构的虚拟筛选
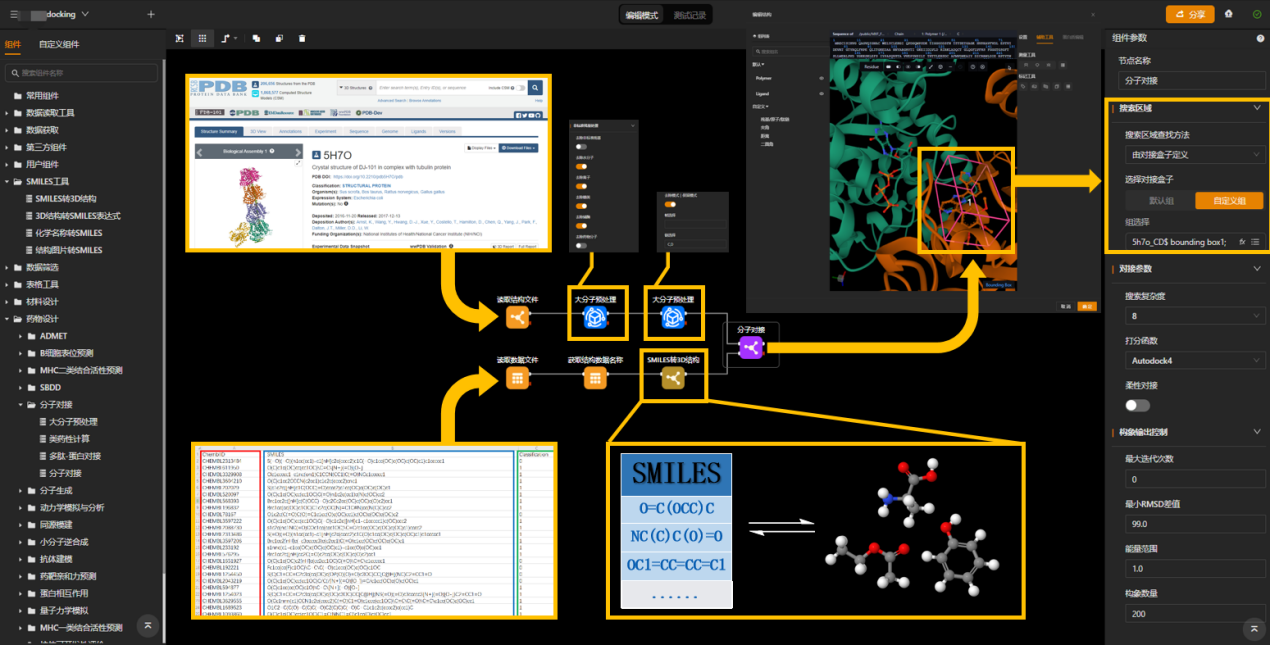
图9.分子对接工作流
计算结果可以显示对接分子的结合模式,打分函数预测的结合亲和力,与打分最佳结果的RMSD值,以及预测的结合模式。根据与关键残基是作用模式和结合亲和力,即可筛选获得预测较好的结构。
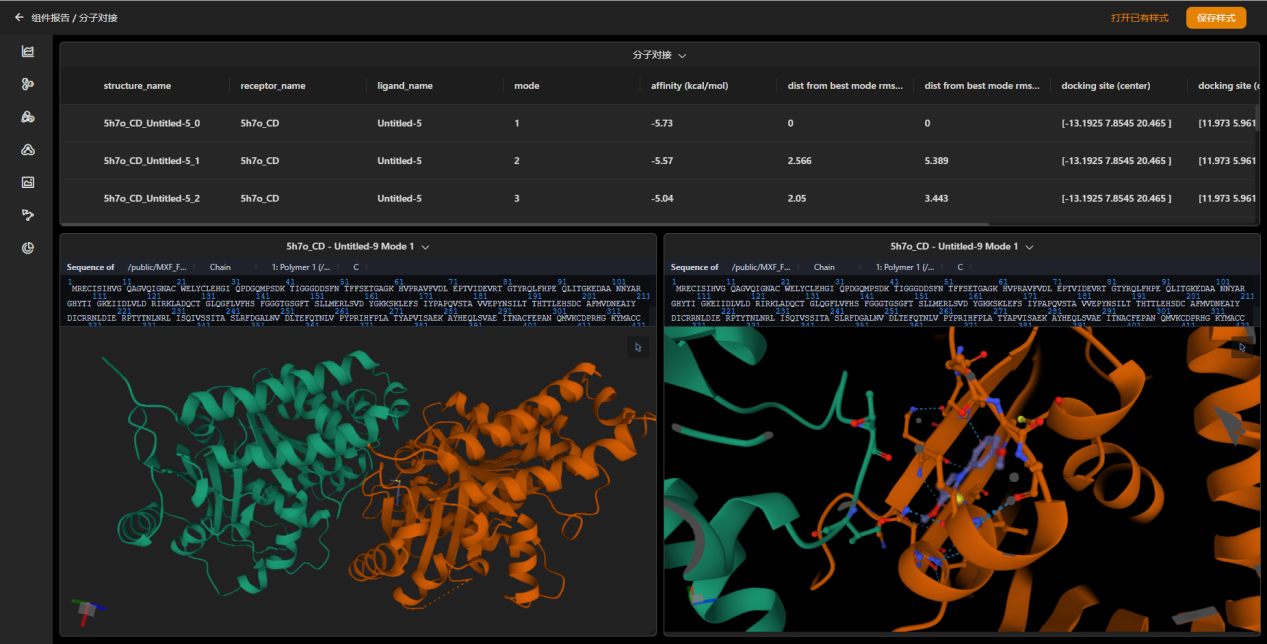
图10.分子对接结果